学院新闻
Small Methods | 赵东宇团队建立了人工智能POCALI方法:预测和理解肿瘤lncRNAs
长非编码RNA(Longnon-coding RNA, lncRNA)作为肿瘤诊断和治疗的新型生物标志物,其研究价值日益凸显,亟需开发生物信息学工具以实现肿瘤lncRNA的系统性识别。现有研究方法多聚焦于基因突变特征,而利用表观基因组等其他组学特征的研究较少。同时,使用突变特征对于识别肿瘤lncRNA是否仍然有效,且不同组学特征对于识别肿瘤lncRNA的贡献程度如何尚不明晰。
针对这一现状,该研究创新性地整合了表观基因组、基因组、转录组、表型、网络和突变六大类共44个多组学特征,开发出具有可解释性的机器学习方法POCALI(图1)。该方法不仅能全面预测肿瘤lncRNA,更能深入解析lncRNA在肿瘤中的潜在作用机制。相关代码已开源发布于https://github.com/starrzy/POCALI。
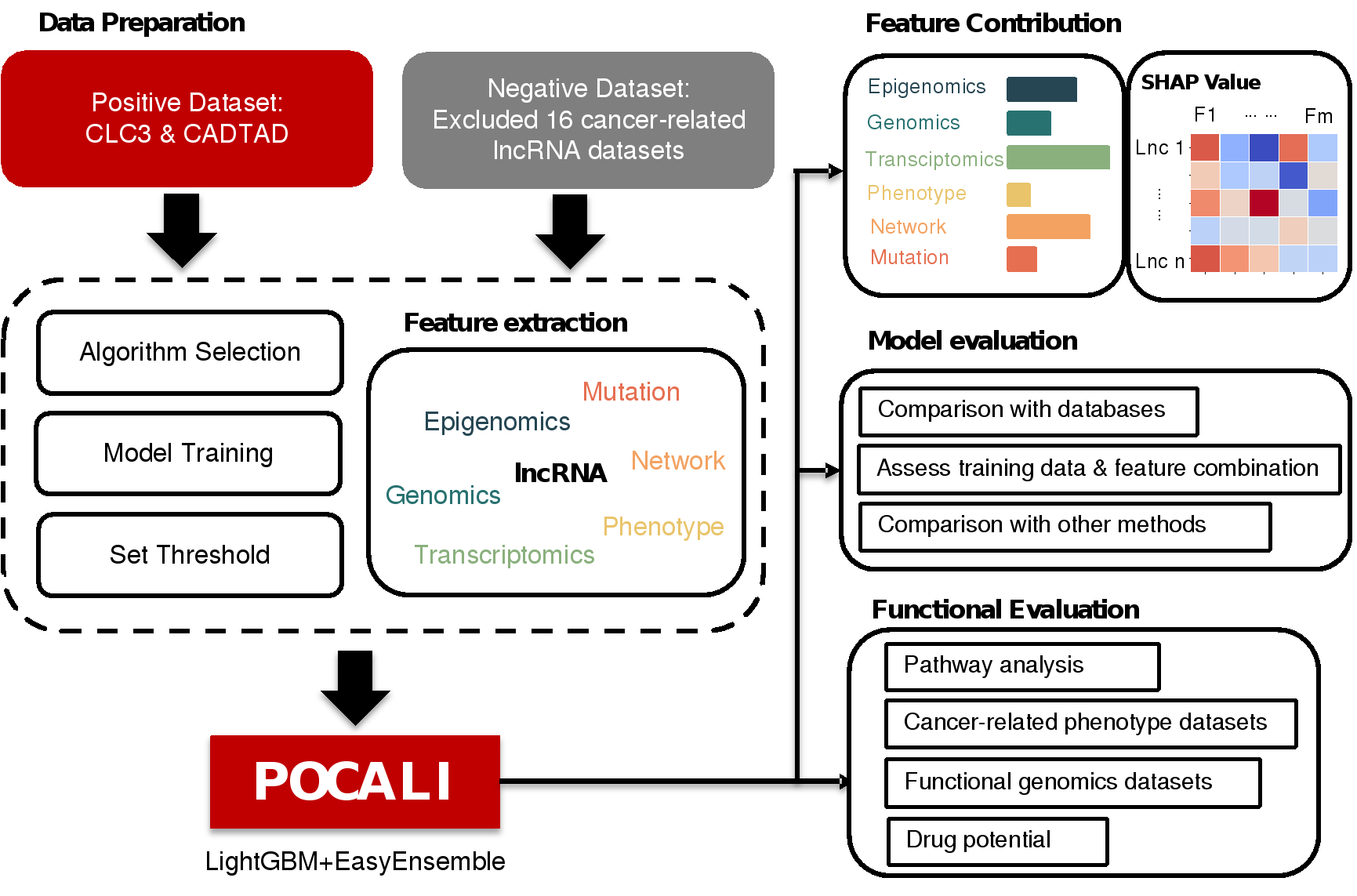
图1POCALI方法流程图
2025年5月23日,北京大学基础医学院医学生物信息学系赵东宇研究员团队在SmallMethods期刊发表了题为“POCALI: Prediction and Insight on CAncerLncRNAs by Integrating Multi-Omics Data with Machine Learning”的研究论文。

POCALI采用LightGBM框架结合EasyEnsemble方法构建预测模型,并创新性地引入SHAP方法实现全局和局部双维度的结果解释。研究发现:二级结构和基因表达相关特征是肿瘤lncRNA的强预测因子,表观基因组特征为中等预测因子,突变特征的预测贡献相对有限(图2)。该方法还可良好地解析每个特征对于预测单个肿瘤lncRNA的贡献程度(图3),并提供在线分析工具(https://huggingface.co/spaces/rzy99/POCALI_feature_analysis),支持用户深入探索特定肿瘤lncRNA的潜在作用机制。与现有方法相比,POCALI展现出更优的性能表现(尤其在敏感性指标上),并成功鉴定出更多潜在肿瘤lncRNA。值得注意的是,这些新发现的肿瘤lncRNA与已知肿瘤lncRNA具有相似的特征谱,均表现出明显的肿瘤相关表型。
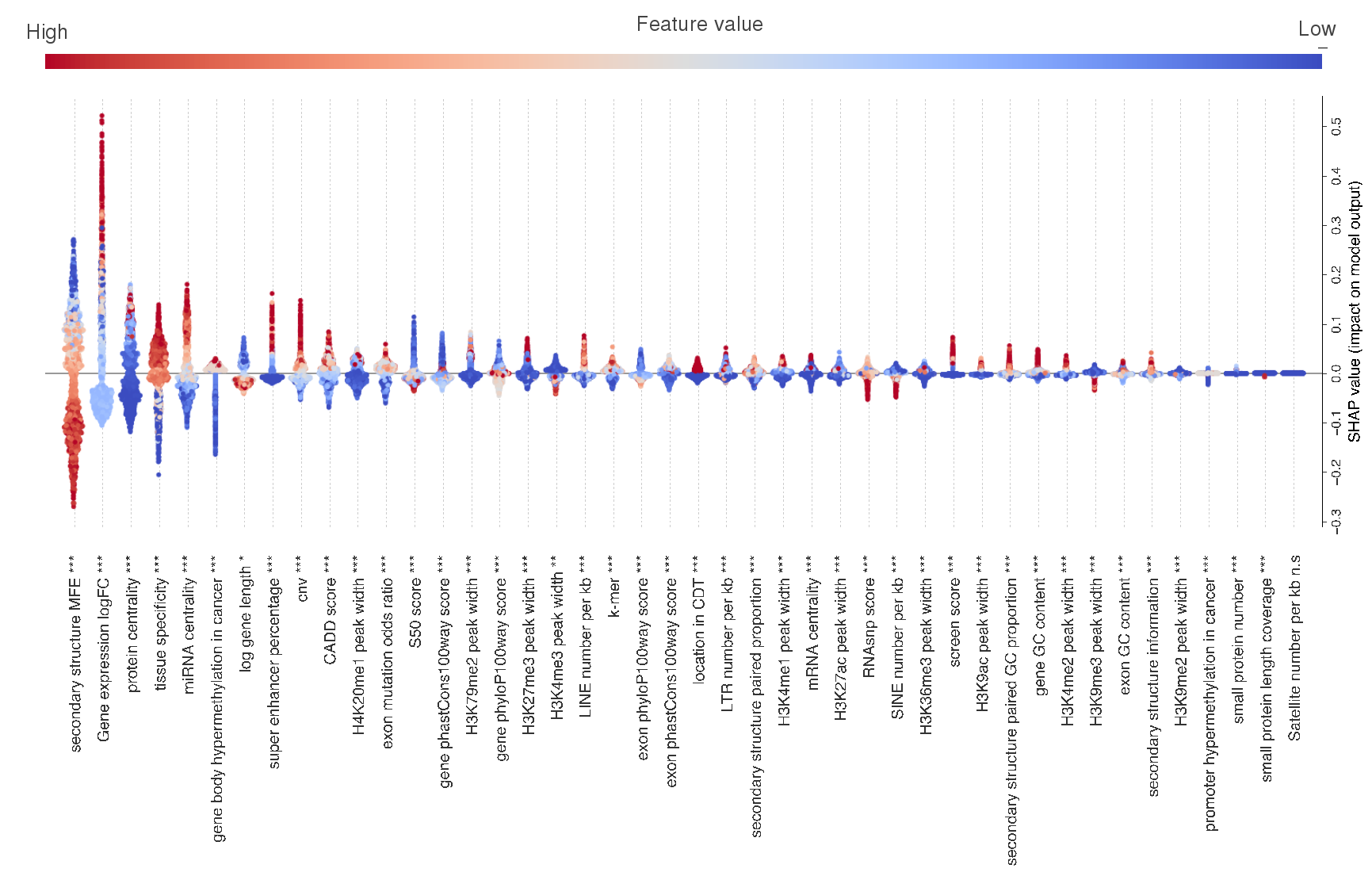
图2全局角度特征重要性分析
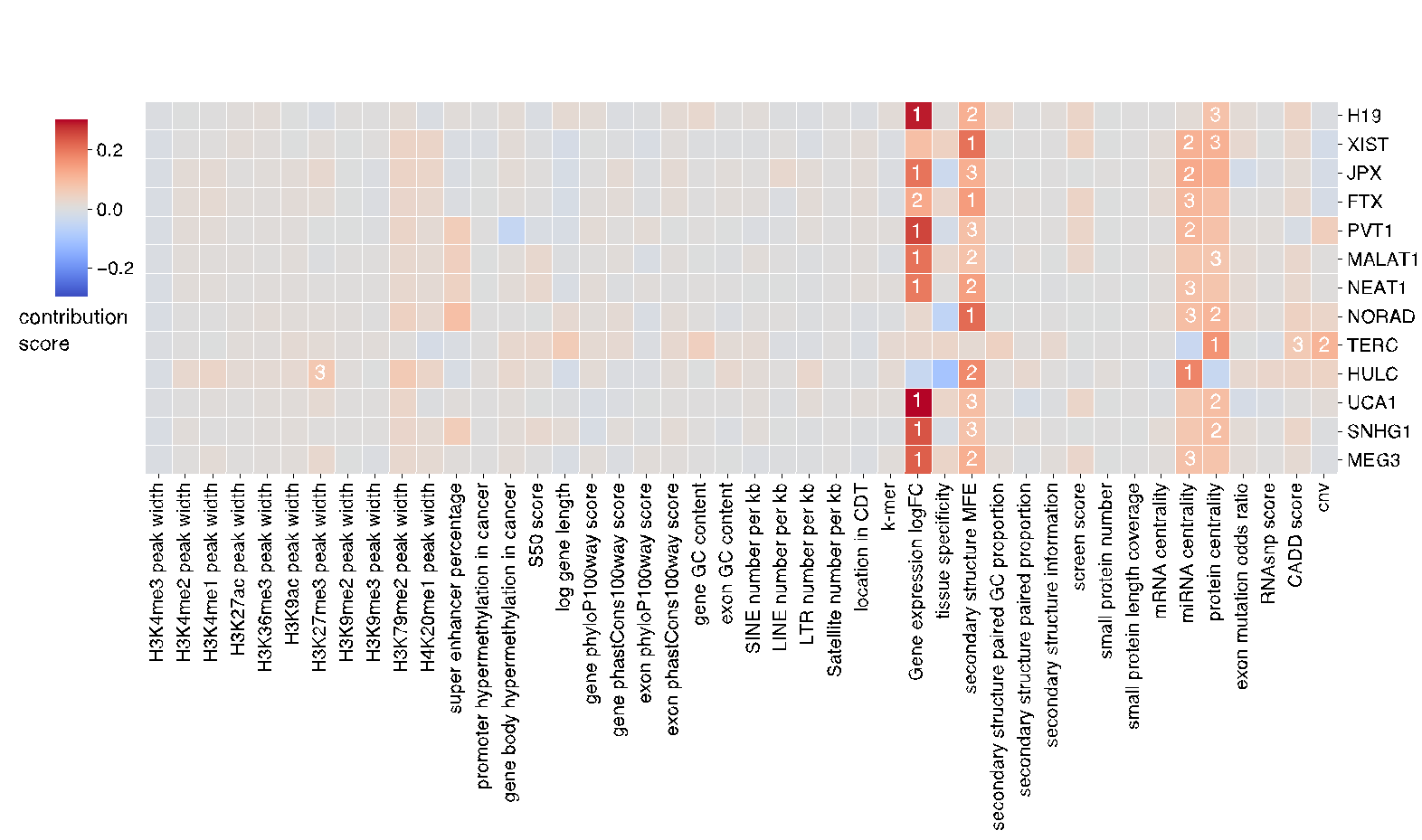
图3局部角度特征重要性分析
综上所述,POCALI作为预测和理解肿瘤lncRNA的新方法,为全面预测肿瘤lncRNA提供了新工具,同时为探究lncRNA在肿瘤中的潜在机制提供了新的角度。通过整合六大类共44个多组学特征,POCALI能够揭示不同组学对于预测肿瘤lncRNA的贡献程度,对于理解lncRNA在肿瘤中的潜在机制具有重要意义,其新发现的肿瘤lncRNA为肿瘤诊疗提供了新的潜在靶点。
北京大学基础医学院医学生物信息学系赵东宇研究员为本文的独立通讯作者,北京大学基础医学院医学生物信息学系博士研究生饶紫嫣为本文的唯一第一作者。本研究获得国家自然科学基金海外优青/面上项目等资助。
论文链接:https://onlinelibrary.wiley.com/doi/10.1002/smtd.202401987

