学院新闻
Advanced Science|周源/李扬团队通过N6-甲基腺苷(m6A)功能读出 的综合分析揭示了调控人类胚胎干细胞分化的新型m6Areader
2026年1月21日,北京大学基础医学院医学生物信息学系周源研究员与细胞生物学系李扬副教授团队合作在《Advanced Science》杂志上在线发表了一项研究成果——“Comprehensive Profiling of N6-methyladnosine (m6A) Readouts Reveals Novel m6A Readers that Regulate Human Embryonic Stem Cell Differentiation”。

m6A甲基化是哺乳动物中最广泛的RNA修饰类型之一,它通过RNA降解、翻译、可变剪接等功能读出(Readout)影响关键靶基因表达,参与多种生理和疾病过程。但当前研究对m6A下游调控效应与机制认知仍存在空白,其调控效应在不同细胞类型中具有高度特异性,仅依靠修饰水平难以准确预测实际功能,调控效应相关的新型m6A Reader也尚待挖掘。
该研究首先选取了五个代表性的细胞系(A549、hESC、HEK293T、JURKAT、HUVEC),分别对METTL3敲降细胞与对照细胞进行时序转录组测序、核糖体测序与超高深度转录组测序,以系统测定五种细胞系在mRNA半衰期、翻译效率与可变剪接三个层面的m6A修饰的功能读出。通过对测序结果的比较分析,发现三个层面的m6A修饰均表现出较强的细胞类型特异性。进一步,通过GLORI技术测定了A549、hESC和JURKAT的定量甲基化谱,结合公共数据,发现mRNA半衰期、翻译效率与可变剪接的变化与m6A修饰水平仅存在弱相关,提示m6A甲基化位点的基因组上下文可能是更为重要的功能读出影响因素。据此研究人员对公共数据库中的RNA结合蛋白(RBP)数据进行了系统的整理与重分析,结合转录本拓扑特征、RBP结合位点特征等m6A甲基化位点上下文特征,建立基于XGBoost算法的m6A功能读出预测模型Reading-m6A,针对不同细胞类型的不同层面的功能读出预测平均AUROC达到0.8(图1)。

图1. m6A甲基化对不同细胞类型mRNA半衰期的调控
该研究对m6A功能读出预测模型Reading-m6A进行了特征分析,预测并验证了4个新型m6A结合蛋白(DDX6、FUBP3、FXR2和L1TD1)。将HyperTRIBE测序应用于DDX6、FUBP3、FXR2和L1TD1蛋白进行进一步验证,结果显示DDX6、FUBP3、FXR2和L1TD1的靶点均显著富集于GLORI发现的m6A位点附近,且四个蛋白的靶基因均显著与m6A修饰靶基因重叠。基于体外纯化的DDX6、FUBP3、FXR2和L1TD1重组蛋白与体外合成的m6A探针,研究人员利用EMSA证实了四个蛋白与m6A的直接结合。mRNA半衰期测序实验提示FUBP3和L1TD1在人胚胎干细胞中具备调控mRNA稳定性的能力(图2)。采用CRISPR/Cas9敲除技术构建FUBP3、FXR2、L1TD1敲除人胚胎干细胞(hESC)系,结合CCK8增殖实验、三胚层分化诱导及流式细胞术、qRT-PCR检测分化标志基因,发现FUBP3、FXR2和L1TD1主要调控人胚胎干细胞的胚层分化而非自我更新,其中FUBP3通过影响NOLC1和CORO1C的mRNA稳定性调控神经分化。综上,该研究通过对功能读出模型的特征分析与多维度的实验验证,鉴定了具有调控人胚胎干细胞早期分化能力的新型m6A reader。
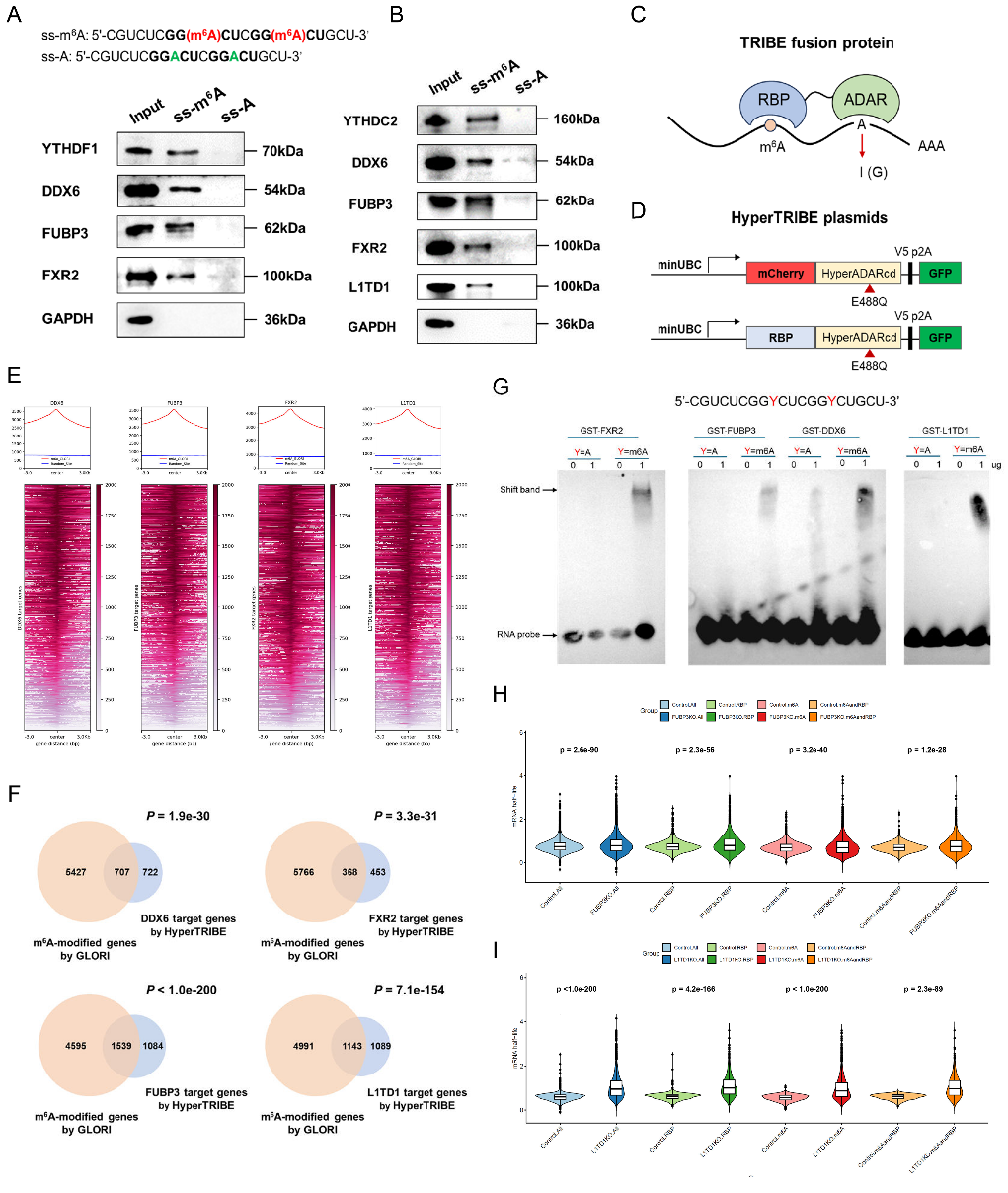
图2. 新型m6A reader蛋白在hESC细胞中的实验验证
该研究结合高通量测序技术与机器学习方法,为全面解析m6A功能图谱提供了新路径,并且系统性建立了m6A功能效应的跨细胞类型分析范式,为解析m6A介导的细胞命运决定机制提供了关键方法与理论依据。
北京大学基础医学院黄洲、刘茹聪、孜巴古丽·吾布力卡斯木与赵婉清为本文的共同第一作者。北京大学基础医学院周源研究员与李扬副教授为本文的通讯作者。北京大学基础医学院崔庆华教授与孔炜教授团队参与了该研究的计算分析与实验验证。该研究得到了国家自然科学基金、北京大学医学部—美国密歇根大学医学院国际合作项目以及北京大学-百度基金项目的资助。
全文链接:https://advanced.onlinelibrary.wiley.com/doi/10.1002/advs.202510075

