学院新闻
周源课题组建立基于图表示深度学习的单细胞转录组数据填补方法GE-Impute
单细胞转录组测序(scRNA-seq)能够从单个细胞水平对转录组进行研究,可以最大程度反映细胞间的异质性,并为鉴定新的细胞亚群和阐明细胞状态转换的调控机制提供技术支持。近年来,10X Genomics、Smart-seq2、Fluidigm C1等技术平台的出现,极大地推动了单细胞测序的发展,使其广泛地应用于基础科研与临床研究中。然而,由于单个细胞的mRNA表达量较低以及测序技术的噪声原因,使得单细胞测序数据中存在较多的drop-out现象,即很多mRNA未被捕捉到导致检测出来基因表达值为零,而这些噪声数据会影响单细胞数据下游分析的准确性。因此,如何准确地填补单细胞数据中的缺失值,并提高下游分析的精确性,是单细胞转录组研究中的一项挑战。
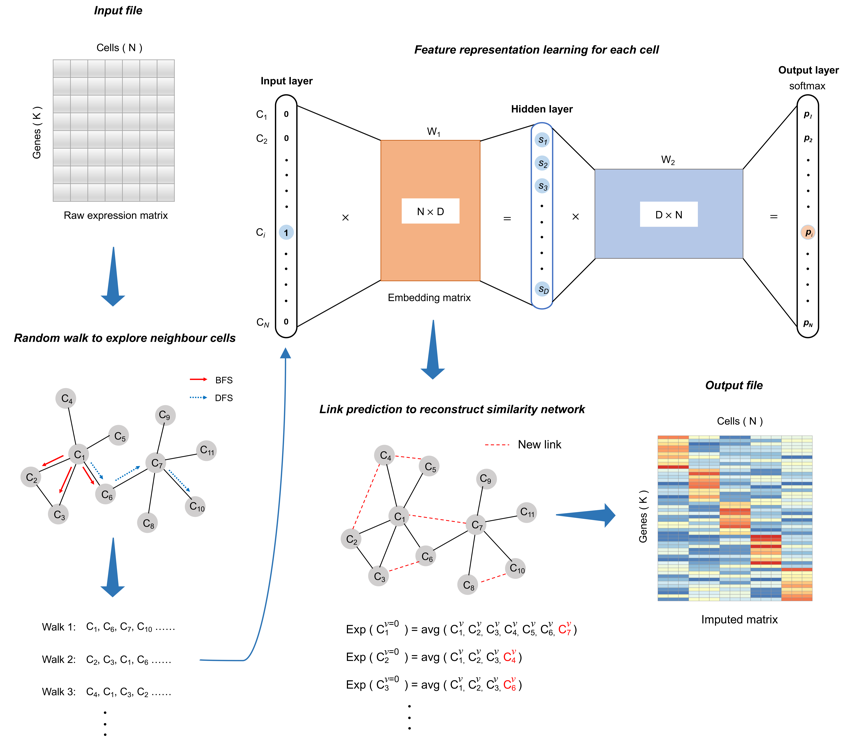
近日,北京大学基础医学院周源课题组在Briefings in Bioinformatics杂志上发表了题为“GE-Impute: graph embedding-based imputation for single-cell RNA-seq data”的研究论文。该方法创新性的将单细胞数据填补表征为相似细胞间链路预测的算法问题,利用图表示深度神经网络学习单个细胞在细胞相似性网络中的关系,并基于这些特征表示预测新的细胞连接并重构细胞相似性网络,从而实现单细胞转录组数据的填补。研究发现,与另外9种主流的单细胞数据填补方法相比, GE-Impute能够更准确地填补来自不同技术平台的(10X Genomics、Smart-seq2、Fluidigm C1)单细胞转录组数据。此外,GE-Impute有效提高了多项下游生物信息分析的准确性,包括细胞类型的聚类、细胞标记基因鉴定与细胞轨迹推断等。
北京大学基础医学院吴晓斌博士为该文章的第一作者,北京大学基础医学院周源副研究员为该文章的通讯作者。该研究得到科技部重点研发计划项目的支持。
周源课题组主要从事转录组、非编码RNA与RNA修饰的生物信息学研究,目前已建立SRAMP、TAM2.0、PACES、m6Acorr、LE-MDCAP等多个生物信息学方法工具。
工具链接:https://github.com/wxbCaterpillar/GE-Impute
原文链接:https://academic.oup.com/bib/advance-article/doi/10.1093/bib/bbac313/6651303
(北京大学基础医学院)

